Hva er fylogeni?
Hvor blir det av septemberbloggen? En doktorgrad er ikke bare å samle data, forske på dem og skrive artikler. Vi må også ta 30 studiepoeng (tilsvarende et halvt års fulltidsstudier) i løpet av doktorgradstiden. Årsaken til at jeg er så treg denne måneden er altså at jeg skal ha eksamen i de siste ti av disse poengene på fredag. Det synes jeg er skummelt, for det er en klassisk skriftlig eksamen og det har gått sju år siden sist jeg gjorde den øvelsen. Ønsk meg lykke til!

Selv om eksamensformen er ubehagelig, er det fint å være student igjen, og et veldig interessant fag: Fylogeni og klassifikasjon. Fylogeni er en stor og viktig del av biologien og paleontologien, men et begrep som kanskje ikke er like godt kjent utenfor den bobla (viser en spørreundersøkelse blant mine familiemedlemmer). Fylogeni er hypotesen om evolusjonen til en gruppe organismer og slektskapet mellom dem, og faget jeg tar nå er et metode-fag. Vitenskap skal være etterprøvbar, og da må vi bruke metoder som er så objektive som mulig. Derfor holder det ikke at jeg ser på ulike fiskeøglearter og skriver noe om hvilke jeg tror var i slekt med hvem basert på magefølelse, det ville aldri bli publisert. Fylogenetisk metode ble utviklet nettopp som svar på subjektive måter å tolke evolusjon og slektskap på, og førte til at vi nå bruker mer matematiske metoder for å analysere hvor like organismer er hverandre.
Likheten finner vi i dataene våre. For nålevende arter brukes stort sett DNA eller andre molekyler, mens jeg ikke har DNA og bruker skjelett og tenner i stedet. Datagrunnlaget deler vi opp i det vi kaller karakterer. Det er en gitt plassering på et molekyl eller et gitt sted på en knokkel, og så legger vi inn i tabellen vår hvordan hver enkelt organisme ser ut. Med DNA sier vi om det er en A, C, G eller T på denne plasseringen, med skjelett for eksempel om fiskeøgla hadde 2 eller 3 underarmsknokler. Molekylære data kan gi tusenvis av karakterer, mens vi for fossiler ofte har noen få hundre.

Så kjører vi denne tabellen igjennom et dataprogram som lager et slektskapstre, og det er reglene vi gir dataprogrammet som er viktige. Hvis treet vi får ut skal si oss noe om evolusjon, må reglene som datamaskinen følger være begrunnet i evolusjonsteori. En metode heter parsimoni og sier at endringer i karakterer er usannsynlige, og dataprogrammet lager et tre der organismene er gruppert slik at så få endringer som mulig har skjedd. I artikkelen vår om fiskeøglene Mikkel og Gypsy brukte vi nettopp parsimoni.
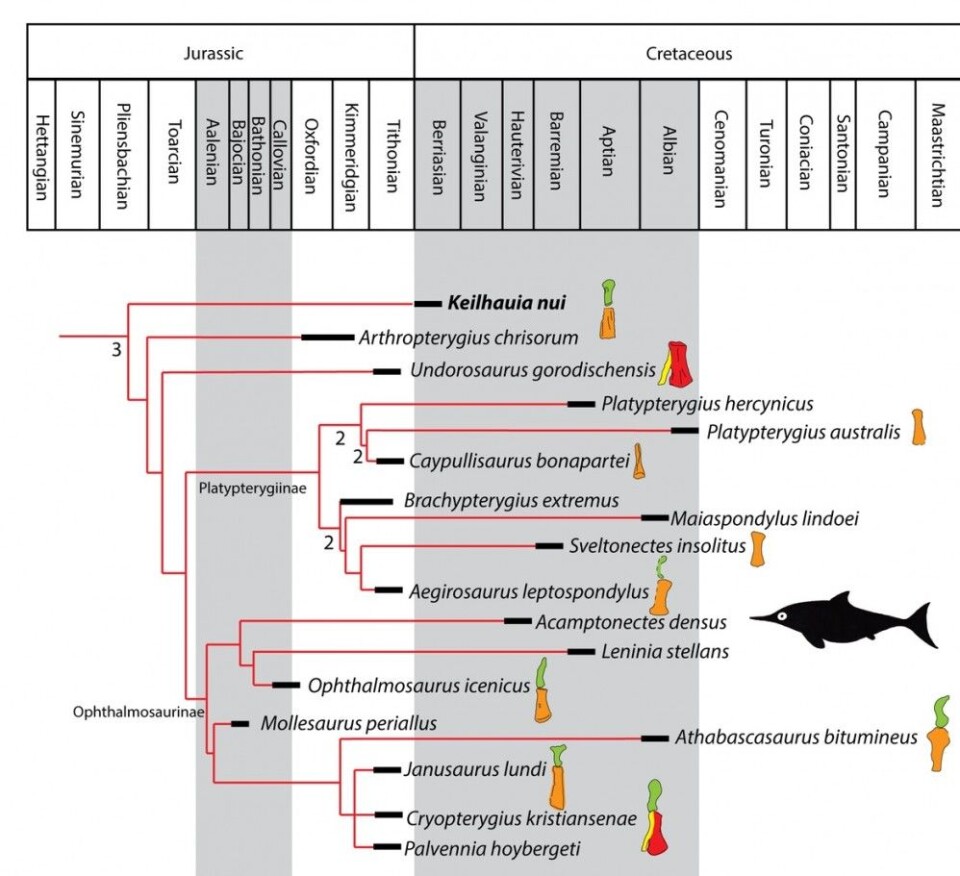
Andre metoder bruker evolusjonære modeller som blant annet handler om hvor sannsynlig det er med ulike typer mutasjoner, og sammenlikner ulike trær for å finne det som passer best med dataene vi puttet inn. Med mange organismer og mange karakterer blir det etter hvert ekstremt mange mulige utseender på trærne, og derfor er fylogenien tett knyttet til utvikling av smarte dataprogrammer og raske maskiner. Allikevel er det i de fleste tilfeller umulig for maskinene å sjekke alle mulige trær, og det er utviklet raffinerte metoder som forhåpentligvis allikevel gir oss et tre som forklarer evolusjonen av dataene våre på en god måte.
Vi kan som regel aldri vite om vi finner det ene sanne treet, men ved hjelp av disse metodene finner vi slektskapstrær som stadig lærer oss mer om naturen og hva det er vi må forske videre på.
Eksamensmodushilsen fra Lene
PS. Det er vanskelig å putte 10 studiepoeng inn på 600 ord, så hvis du ble nysgjerrig kan du for eksempel lese mer om hvordan man forstår slektskapstrær med eksempelet dinosaurer i en tekst jeg skrev på kollokvium.no.







